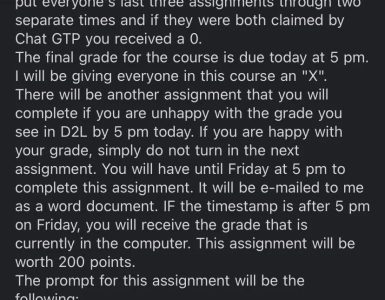
하버드위스연구소 연구팀이 수백만 유전자 실험을 동시에 실시할 수 있는 새로운 유전자 편집 도구를 개발했다. 단일 가닥 DNA 조각을 일으키는 레트론이라는 박테리아 DNA 세그먼트를 조작하는 레트론 라이브러리 재조합 RLR(Retron Library Recombineering)이라는 기술을 이용하는 것.
유전자 편집이라고 하면 지난 몇 년간 크리스퍼(CRISPR-Cas9) 기술이 화제였다. 기존 기술보다 정밀하게 DNA 서열을 쉽게 배치할 수 있어 주로 질병 치료를 위한 연구에 사용되고 있다. 하지만 크리스퍼 기술은 돌연변이 서열을 게놈에 통합하는 목표로 DNA 일부를 절단한다. 이 때 분자 수준 Cas9 효소가 종종 목표와 다른 장소를 잘라 버리기 때문에 세포 수준에서 유해한 것으로 보이는 가능성이 있을 수 있다.
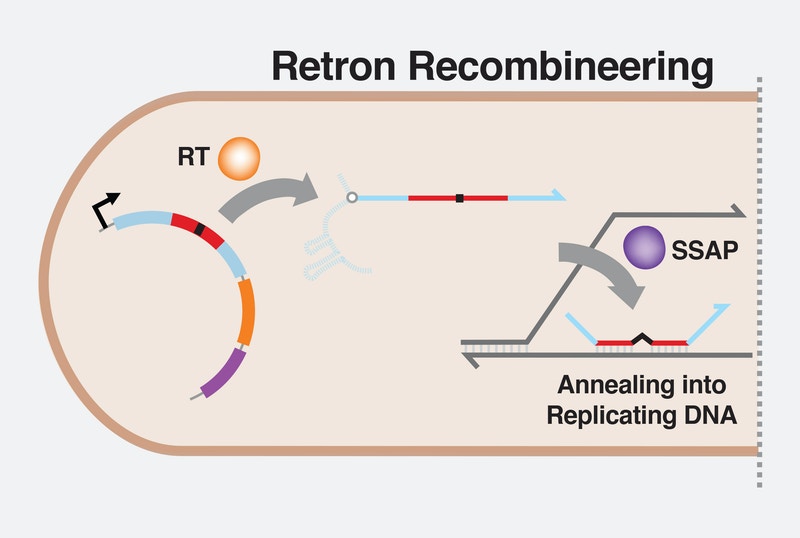
이에 비해 레트론은 변이된 DNA 가닥을 복제 중인 세포에 도입하고 딸세포에 이를 채울 수 있다. 채워진 배열은 분열할 때마다 복제되어 배열 자체가 어떤 개체가 각 레트론 배열을 받았는지 확인하는 바코드 역할을 한다. 이를 통해 정확하게 만들어진 돌연변이주를 추적하기 쉬워진다. 이 방법은 원래 DNA에 손상을 주지 않고 게놈 편집을 한 번에 대량 배열을 투입해 여러 실험을 실행할 수 있게 해준다.
연구팀은 대장균에서 RLR을 실험하고 몇 가지 조정을 실시한 결과 90% 개체가 레트론 배열을 넣을 수 있게 되는 걸 확인했다. 실험에선 개별 돌연변이 염기 서열을 알아내는 대신 레트론 바코드 염기 서열을 조사해 대장균 항생제 내성 변이를 찾는 과정을 대폭 단축할 수 있었다.
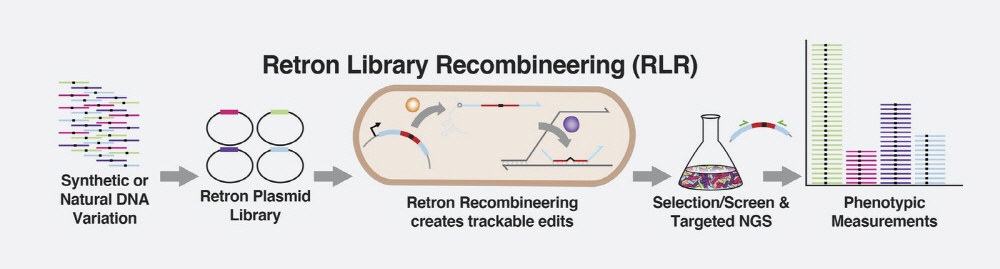
연구팀은 RLR을 한 걸음 더 나아가 무작위로 조각난 DNA에 이 기술을 적용할 수 있는지, 또 한 번 사용할 수 있는 레트론 수를 조사했다. 이들은 다른 항생제에 내성을 가진 대장균 게놈을 재단하고 이 조각을 이용해 플라스미드라는 딸 세포에 분배되는 염색체 외에 DNA 분자 레트론 배열에 포함된 수천만 유전자 배열 라이브러리를 구축했다.
연구팀은 RLR을 이용해 풀링된 바코드 돌연변이 라이브러리를 분석할 수 있게 해 수백만 실험을 동시에 수행할 수 있다면서 이제 게놈 전체 변이 영향과 해당 변이가 어떻게 상호 작용하는지 조사하는 것도 가능하게 됐다고 밝혔다. 또 이 연구는 RLR이 다른 유전자 시스템에서 사용되기 위한 로드맵을 수립하는데 도움이 되고 앞으로 유전자 연구에 흥미로운 가능성을 제공하게 될 것이라고 밝혔다. 관련 내용은 이곳에서 확인할 수 있다.