
딥마인드가 유전자 서열 정보로부터 단백질 입체 구조를 예측하는 기술인 알파폴드(AlphaFold)를 개발했다. 정확한 단백질 입체 구조를 이해하게 된다면 알츠하이머나 파킨슨 등에 대한 신약 개발에도 도움이 될 것으로 기대를 모으고 있다.
단백질은 근육을 수축시키는 빛을 감지하는 음식을 에너지로 바꿔주는 등 인간의 몸이 할 수 있는 거의 모든 기능과 관련이 있다. 단백질은 20종 가량 존재하는 L-아미노산이 사슬 모양으로 다수 연결된 고분자 화합물로 아미노산 잔기로 불리는 아미노산 단위가 어떻게 연결되어 있는지는 1차원적인 배열 정보로 DNA에 기록되어 있지 않다. 따라서 단백질의 입체 구조를 예측하는 건 단백질 접힘(Protein folding) 문제라는 과제가 되고 있다. 단백질 접힘이란 선형으로 이뤄진 아미노산 복합체인 단백질이 개개 단백질에 고유한 접힌 구조를 만드는 과정을 말한다. 어쨌든 이런 문제로 단백질이 클수록 모델링은 복잡하고 어려워지는 것으로 알려져 있다.

지금까지 연구에선 저온 전자현미경법이나 핵자기공명, 엑스선 결정학을 통해 단백질의 입체 구조를 밝혀왔다. 하지만 어떤 방법도 구조마다 시행착오가 있고 비용이 많이 드는 게 문제였다. 이런 이유로 양자택일 방법으로 인공지능을 이용한 예측이 주목받게 된 것이다.
지난 몇 년 사이 유전자 분석 기술이 크게 진화하면서 유전자 분야 데이터를 풍부하게 준비할 수 있게 됐고 딥마인드 역시 이런 데이터를 바탕으로 알파폴드를 개발하는 데 성공하게 된 것.
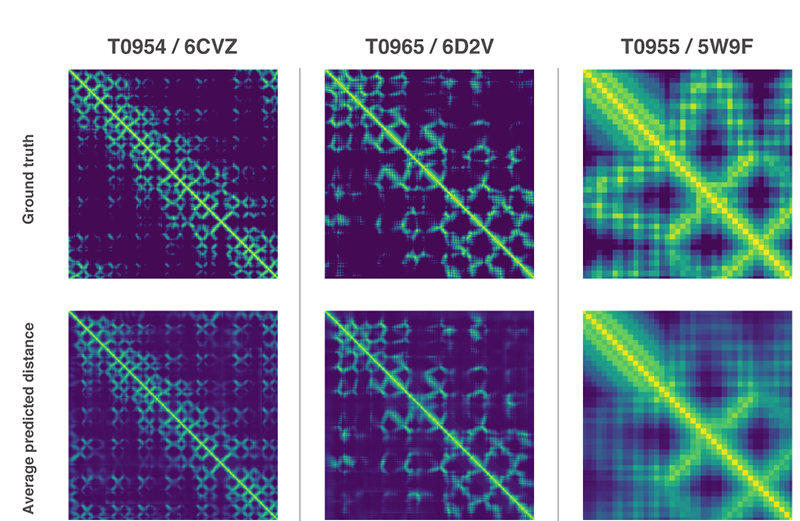
연구팀은 과거 해결된 단백질 접힘 문제를 이용하는 게 아니라 대상 구조 모델링을 처음부터 다시 착수했다. 단백질 구조를 가진 물질 특성을 예측한 경우 예측 정확성이 높아지는 것으로 판명된 만큼 연구팀은 2가지 방법을 이용해 전체 단백질 구조 예측을 실시했다.
2가지 방법은 모두 유전자 배열에서 단백질 특성을 예측하도록 훈련한 신경망을 이용한다. 네트워크가 예측하는 특성은 아미노산 쌍의 거리와 그들을 연결하는 화학 결합 사이의 각도 2가지다. 이 2가지 예측에서 얼마나 정확한 단백질 구조를 제시할 수 있는지를 점수화하고 이 점수는 머신러닝을 이용해 기울기하강법(Gradient descent, steepest descent)에 최적화, 높은 정확도로 예측이 가능하게 됐다고 한다.
앞서 밝혔듯 알츠하이머나 파킨슨, 헌팅턴, 낭포성 섬유증 같은 질병의 원인은 단백질의 잘못된 접힘이 원인인 것으로 추정되고 있다. 단백질 구조를 3차원적으로 예측하고 이해하는 건 새로운 약물 개발에도 도움이 될 것으로 생각되는 이유다. 또 단백질 접힘을 이해하면 생분해성 효소 분야에서도 진보를 기대할 수 있어 플라스틱이나 기름 등 폐기물 분해 역시 더 환경친화적인 형태로 이뤄지게 될 가능성이 있다. 관련 내용은 이곳에서 확인할 수 있다.

