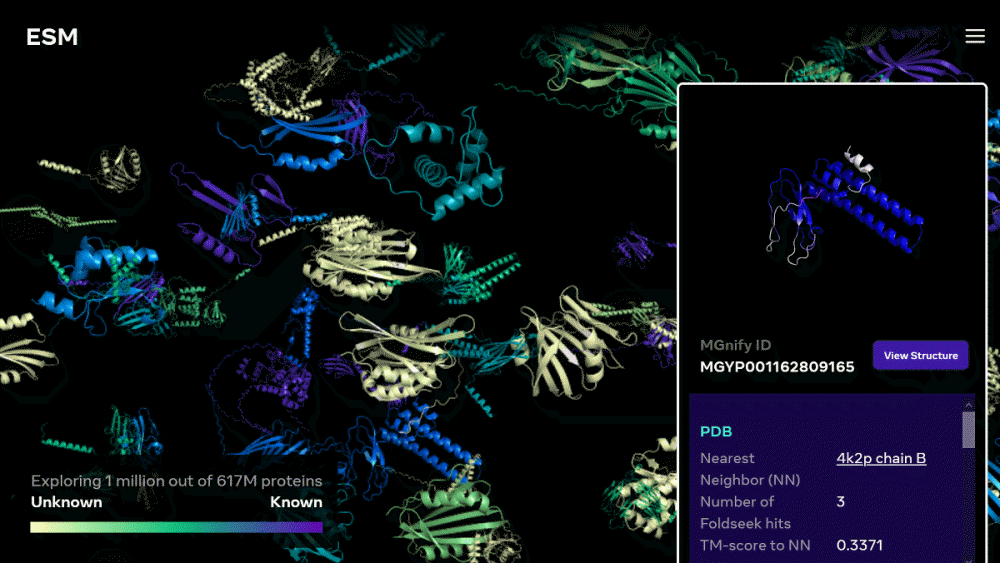
메타가 단백질 입체 구조를 예측하는 언어 모델인 ESM-2를 이용해 6억 1,700만 종 이상 메타 게놈 단백질 구조를 예측한 데이터베이스인 ESM 메타게노믹 아틀라스(ESM Metagenomic Atlas)를 공개했다.
생물을 구성하는 단백질에 대해 아는 건 생물학과 의학 연구에서 중요하다. 하지만 아미노산끼리 결합한 폴리펩티드 사슬이 접힌 단백질 입체 구조를 예측하는 건 단백질 접힘(protein folding)으로 불리며 연구자에게 있어 어려운 과제가 되고 있다.
단백질 접힘 문제에 대처하는 수법으로 최근에는 AI를 이용해 단백질 입체 구조를 예측하려는 시도가 진전되고 있다. 알파벳 산하 AI 기업인 딥마인드가 개발한 알파폴드(AlphaFold)라는 단백질 구조 해석 AI는 단시간에 저비용으로 실험적 기법과 동등한 정밀도로 단백질 입체 구조를 해석할 수 있게 했다. 알파폴드는 2021년 7월 오픈소스화되어 생물학 세계를 변화시킨 것으로 평가되고 있다.
2022년 7월에는 알파폴드가 예측한 2억 종 이상 단백질 입체 구조가 검색 가능한 데이터베이스로 공개됐다. 이어 11월 페이스북 등을 운영하는 메타 AI 연구팀이 6억 1,700만 종 이상 메타게놈 단백질 구조를 예측한 데이터베이스인 ESM 메타게노믹 아틀라스를 공개한 것. 메타게노믹스는 환경 샘플에서 직접 회수된 게놈을 다루는 연구 분야로 메타는 메타게놈 시퀀스를 카탈로그화하는 공개 자원 MGnify90에 포함되는 게놈 단백질 구조를 예측했다고 한다.
연구팀은 자신들이 아는 한 ESM 메타게노믹 아틀라스는 고해상도 예측 단백질 구조를 모은 가장 큰 데이터베이스라며 대규모로 다루는 첫 데이터베이스이기도 하다고 밝혔다. 또 이 단백질 구조는 자연 넓이와 다양성에 대한 전례없는 견해와 새로운 과학적 통찰력을 제공하며 의료와 자연 화학, 환경 응용, 재생 가능 에너지 등 분야에서 실용화되는 단백질 발견을 가속화할 가능성이 있다고 밝혔다.
메타의 단백질 구조 예측 AI는 ESM폴드(ESMFold)로 명명됐으며 단백질을 구성하는 원자와 분자를 언어로 해석하고 학습 데이터로부터 입체 구조를 예측하는 모델이다. 연구팀은 이 모델을 확장하고 150억 개 파라미터를 가진 ESM-2를 개발했다. ESM-2은 지금까지 가장 큰 단백질 언어 모델이며 ESM 메타게노믹 아틀라스에 포함된 6억 종 이상 단백질 입체 구조를 2,000개 GPU를 이용해 불과 2주 만에 예측할 수 있었다고 보고하고 있다.
연구팀에 따르면 ESM-2 예측 정밀도는 알파폴드만큼은 아니지만 구조 예측 속도는 무려 60배에 달한다고 한다. 메타 측은 이게 의미하는 건 구조 예측을 훨씬 더 큰 데이터베이스로 확장할 수 있다는 것이라고 말한다. 관련 내용은 이곳에서 확인할 수 있다.


